IGV快速上手指南(三)——查看突变信息
当我们将BAM文件导入IGV时,会发现BAM的read track中有很多特殊的符号或颜色,这些特殊符号或颜色可以为发生的突变提供依据。一般情况下,会先用专门的工具分析出突变的位置和类型,再根据分析结果在IGV中进行查看。
特殊符号
插入突变(Insertions)

插入突变会以紫色的"I"展示,中间的白色数字是插入的碱基数,点击插入突变的符号后会弹出具体插入的碱基。

缺失突变(Deletions)

缺失突变会以黑色"—"的形式展示,中间的紫色数字是缺失的碱基数。

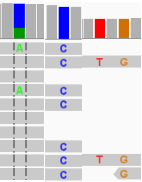
点突变(SNP)

点突变会以实际的碱基进行着色,放大后会显示具体的碱基。默认情况下,绿色为A,红色为T,蓝色为C,黄色为G。Coverage track中也会根据原始碱基和突变碱基的数量显示相应比例的色块。
read颜色
在BAM文件中,Reads的颜色根据选择的模式(在BAM的TRACK中右键选择"Color alignments by",再选择特定的模式)会有所变化。其中,insert size、pair orientation和insert size and pair orientation是查看突变时最常用的三个模式。

insert size

reads根据插入片段长度着色,可用于推断结构突变(SV)——插入、缺失和染色体间重排。

-
灰色表示insert size正常,是常规reads。
-
红色表示insert size大于预期,表示可能存在缺失突变。
-
蓝色表示insert size小于预期,表示可能存在插入突变。
如果reads显示灰色、红色、蓝色以外的其他颜色,表示paired end reads中成对两条reads比对到了不同的染色体。不同颜色表示不同的染色体,是可能存在染色体间重排的依据。如果看到特殊颜色的reads,且想不起来对应的颜色表示比对到什么染色体上,可以点击这条read,在弹出窗口中可以看到成对的另一条reads比对到的位置,如下图。

pair orientation

reads根据pair-end reads的方向着色,可用于推断结构变异——倒置、重复和易位。

IGV展示的reads信息中有个Pair orientation,和这里的着色对应。

1和2分别表示read1和read2,F和R分别表示比对到正链(forward strand)和负链(reverse strand)。
-
LR:正常reads,reads方向为F1-R2或F2-R1,即两条reads比对到基因组后头对头;
-
LL:倒置(Inversions)或反向复制(Inverted Duplication)的reads,reads方向为F1-F2或F2-F1,即两条reads比对到基因组后方向相同,且都在正链上;
-
RR:倒置(Inversions)或反向复制(Inverted Duplication)的reads,reads方向为R1-R2或R2-R1,即两条reads比对到基因组后方向相同,且都在负链上;
-
RL:串联重复(Tandem Duplication)或易位(Translocation on the Same Chromosome)的reads,reads方向为R2-F1或R1-F2,即两条reads比对到基因组后尾对尾。
|
LL/RR |
Inversions倒置 |
 |
|
Inverted Duplication反向复制 |
 |
|
|
RL |
Tandem Duplication串联重复 |
 |
|
Translocation on the Same Chromosome易位 |
 |
insert size and pair orientation

同时根据insert size和paired-end reads的方向着色,着色规则与各自的规则相同。如果reads同时有insert size和方向的异常,会优先根据方向着色。一般情况下,如果需要查看所有类型的结构变异,可以直接按照insert size and pair orientation模式查看。
下期预告:
下一节我们将会演示BAM文件中融合基因的查看。

往/期/回/顾










