IGV快速上手指南——(一)安装和使用
1
简介
IGV(Integrative Genomics Viewer基因组浏览器)是一个将基因组学数据可视化的可交互工具,具有图形界面,高性能且易于使用。可用于展示和查看基因表达、突变、融合、甲基化、探针等多种信息的软件,是展示NGS数据和分析结果的一项重要工具。
2
安装
IGV下载链接:https://software.broadinstitute.org/software/igv/download
根据电脑环境选择MacOS/Windows/Linux版本进行安装,如果不确定电脑是否已经安装Java 11,可以直接安装Java included的版本。
3
使用
以查看BAM文件为例,简单地演示一下使用方式。(以windows 2.13.0版本为例):
图文教程:
01
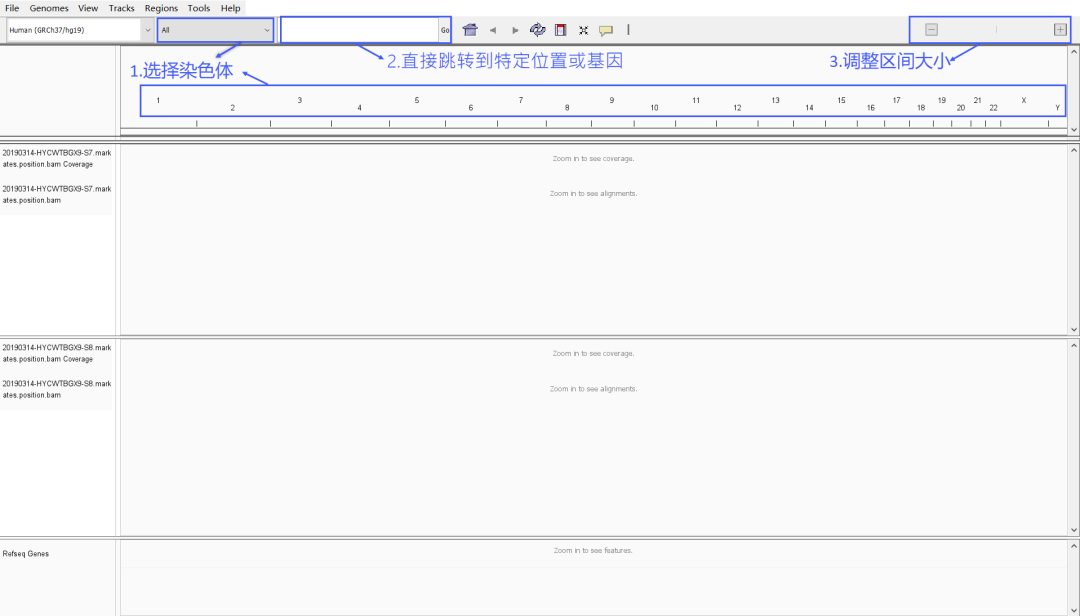
选择基因组版本
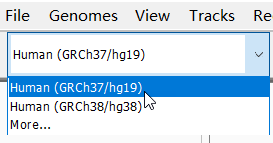
在工具栏的第一个下拉框中可以选择基因组版本,因为分析比对使用的基因组版本为人的GRCh37版本,所以这里选择Human(GRCh37/hg19)。
02
导入BAM文件
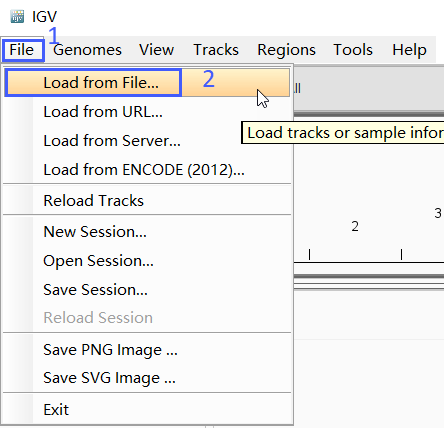
在菜单栏中选择"File" "Load from File…"可以导入本地文件,这里选择index后的*.bam文件。
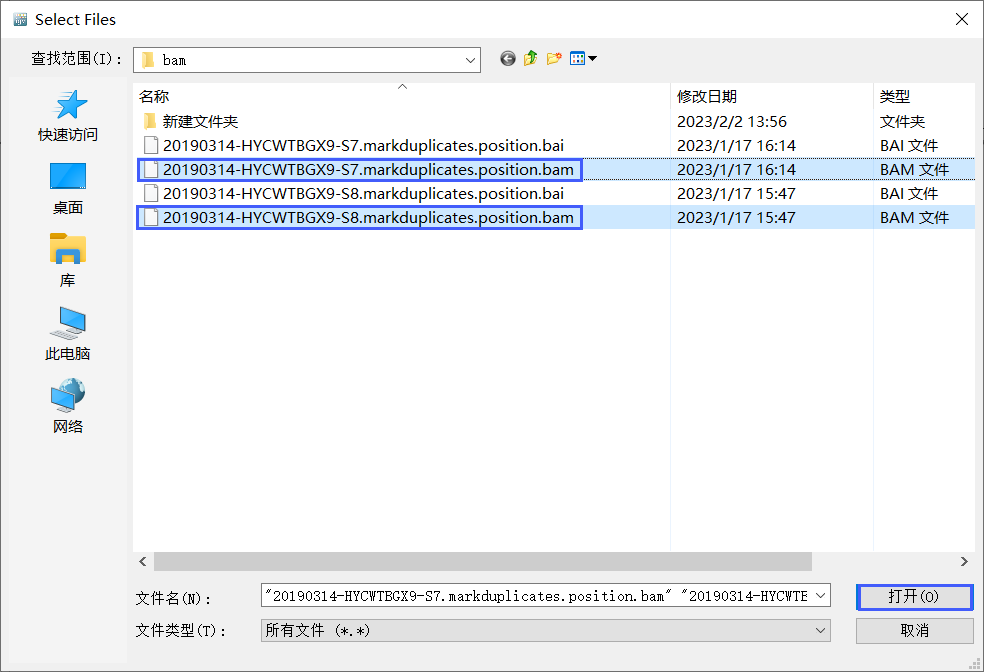
请注意:*.bam所在文件夹下最好有对应的*.bai文件。如果同路径下没有*.bai文件,直接导入bam文件,会弹出一个窗口要求设置bai文件的所在路径,如下图:
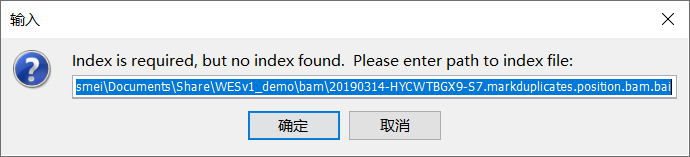
如果选择的路径下仍没有*.bai文件,会弹出报错信息,Index file not found:

假如确实没有对应的bai文件,可以利用IGV中的igvtools生成bai文件。但是请注意,使用igvtools生成bai文件后,导入bam文件时需要选择生成的sorted bam而不是原始的bam文件。方法如下:
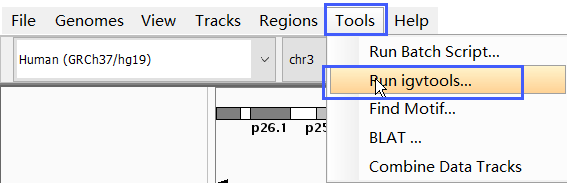
a
在菜单栏选择"Tools""Run igvtools…"
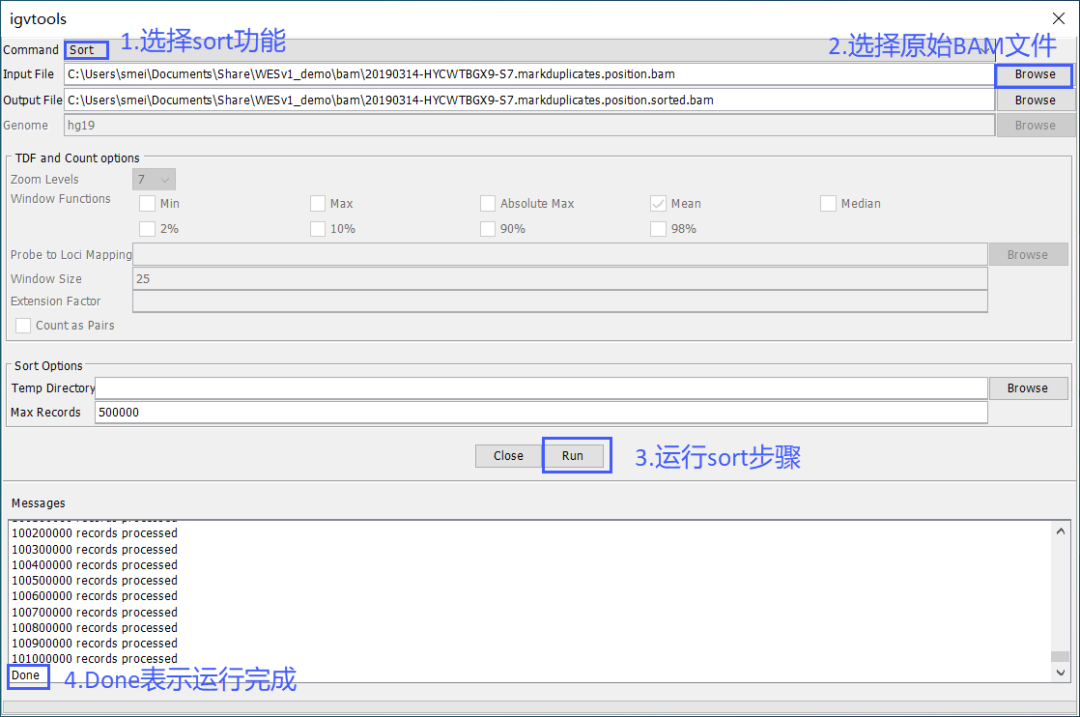
b
在igvtools的窗口选择sort功能,导入原始的bam文件,将原始的BAM进行排序。运行完成后会生成一个sorted bam文件。
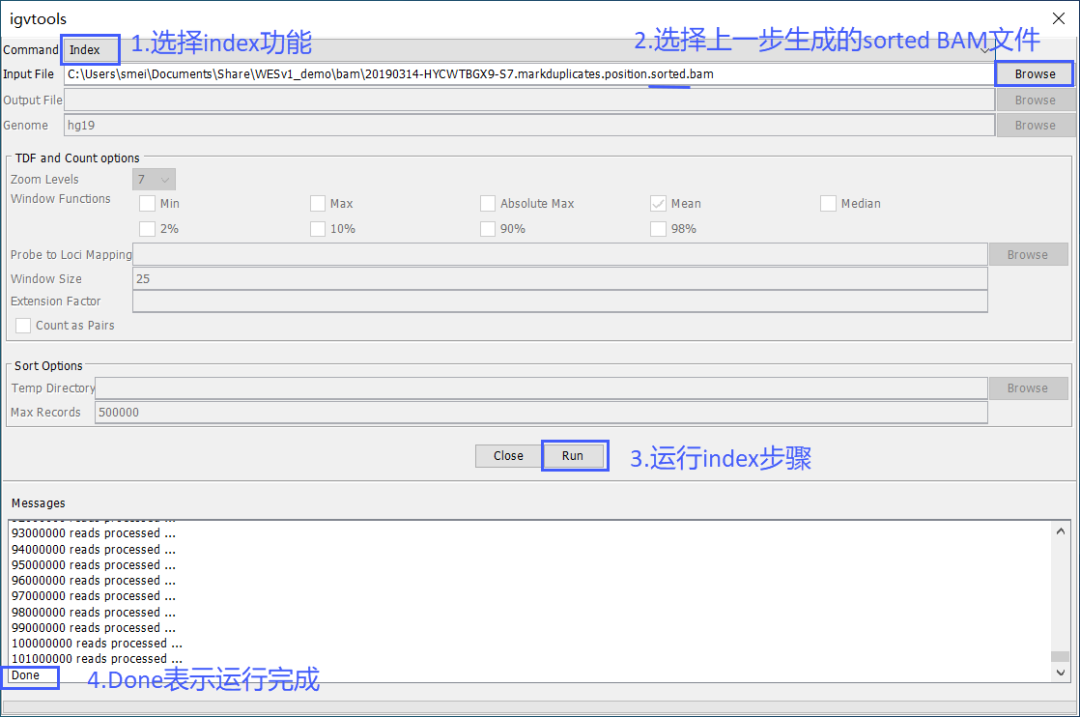
c
等sort完成后,选择index功能,再导入上一步生成的sorted bam文件,建立索引,生成bai文件。
03
选择染色体/跳转到感兴趣的位置
文件导入后显示的是全基因组的状态。在染色体下拉框中选择或者直接点击下方的特定染色体都可以跳转到对应的染色体试图中。在搜索框中输入具体位置区间(如chr1:32134658-32163866或chr1:32162541)或基因名(如AKT3)后点击"Go"或敲击回车键就可以跳转到对应位置。
4
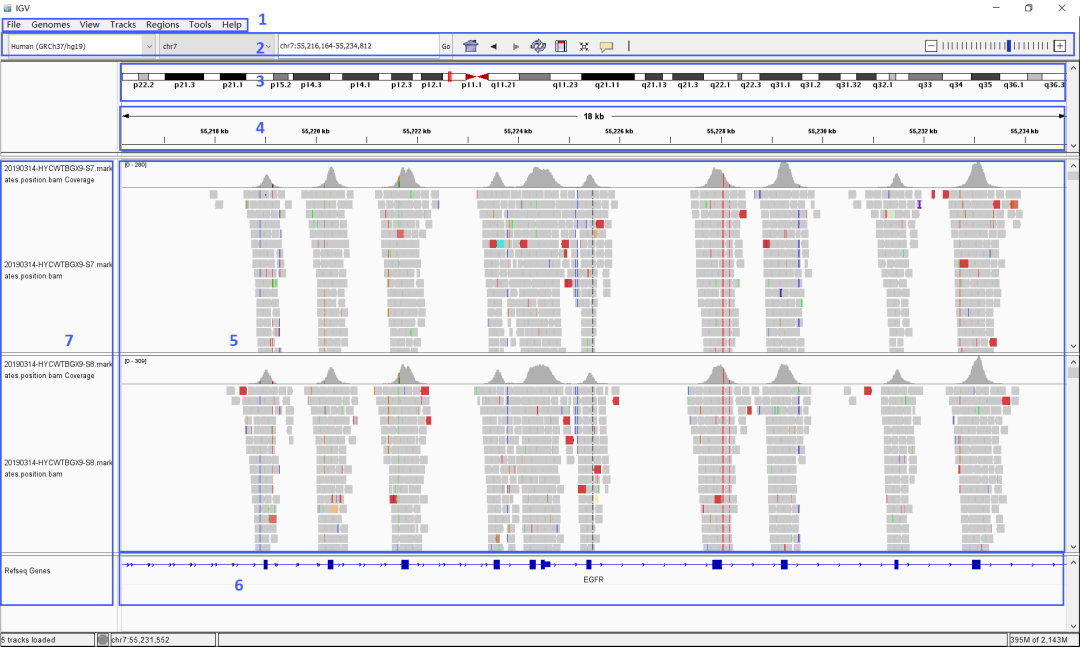
主界面介绍
-
菜单栏,可以进行各种功能的选择。
-
工具栏,列出了常用的几个功能。
-
染色体位置,染色体上的红框表示目前正在展示的位置。
-
展示区间在染色体上的具体位置,上方的跨度表示区间的大小。
-
导入的数据信息,每一行都叫做一个track。因为这里导入的是BAM文件,因此每个样本会有两个track,上面的track展示coverage情况,下面的track展示具体的reads信息。
-
Feature注释信息,默认展示基因转录本信息,可以额外添加探针信息、SNP位置等信息。
-
每个track的名称。
下期预告:
下期我们将会进行实战操作,演示如何在IGV中结合bed文件查看探针捕获数据,敬请期待。



扫一扫 关注我们
上海仁科生物科技有限公司
医学和生命科学前沿研究完整解决方案供应商








